matlab中的分水岭算法
anyone knows how to write a function in matlab to segment the cells and compute the average
cell area using the watershed algorithm?
any help would be much appreciated. Thank you!
Here is an image of yeast cells
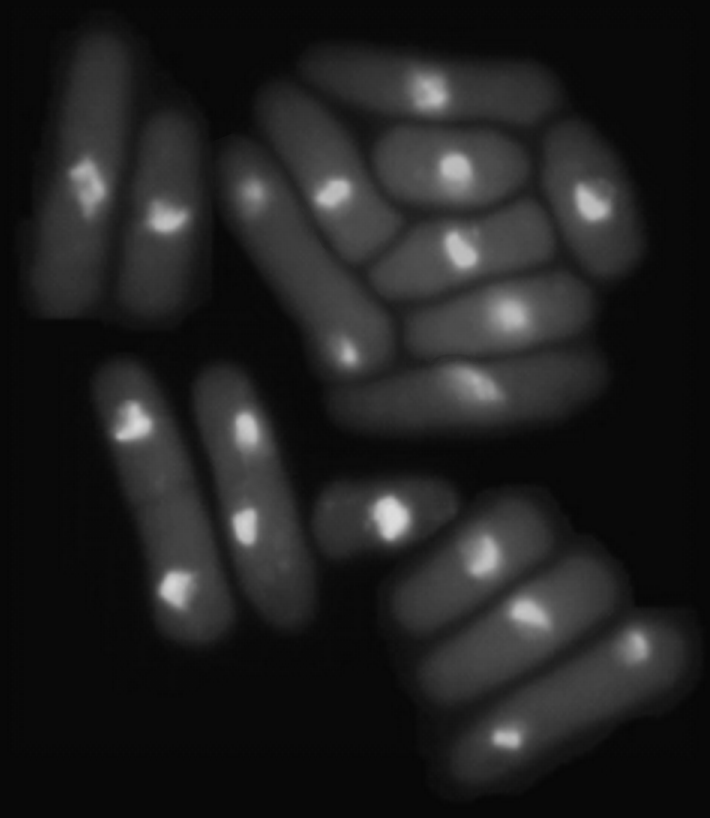
如果你对这篇内容有疑问,欢迎到本站社区发帖提问 参与讨论,获取更多帮助,或者扫码二维码加入 Web 技术交流群。

绑定邮箱获取回复消息
由于您还没有绑定你的真实邮箱,如果其他用户或者作者回复了您的评论,将不能在第一时间通知您!

发布评论
评论(2)
这是使用分水岭分割图像的一种方法。您还可以做更多的事情(例如,如果细胞尚未完成胞质分裂,则融合具有两个细胞核的细胞),但下面的步骤应该会给您一个初步的想法。
(1) 确定细胞背景阈值、细胞核阈值
(2) 平滑原始图像(以避免过度分割)并将细胞核强加为最小值
(3) 分水岭、遮罩细胞并显示
(4) 现在您可以使用 REGIONPROPS 在标记图像上提取所需的统计信息。
Here's one way to segment the image using watershed. There's plenty more you could do (e.g. fuse cells with two nuclei if they haven't completed cytokinesis yet), but the steps below should give you a first idea.
(1) Determine cell-background threshold, cell-nucleus threshold
(2) Smooth the raw image (to avoid oversegmentation) and impose nuclei as minima
(3) Watershed, mask cells, and display
(4) Now you can use REGIONPROPS on the labeled image to extract the statistics you want.
请参阅图像处理中的
watershed工具箱和关于细胞分割的这篇文章图像处理博客。See
watershedin the Image Processing toolbox and this post on cell segmentation on the 'Steve on Image Processing' blog.